
图1 Molecular Plant在线发表研究论文BnVIR: bridging the genotype-phenotype gap to accelerate mining of candidate variations for traits in Brassica napus
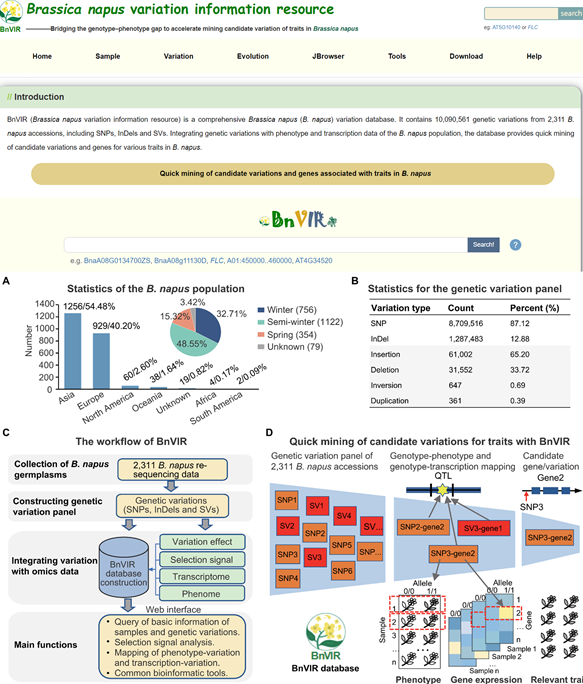
图2 BnVIR数据库主页、构建流程和概貌(http://yanglab.hzau.edu.cn/BnVIR)
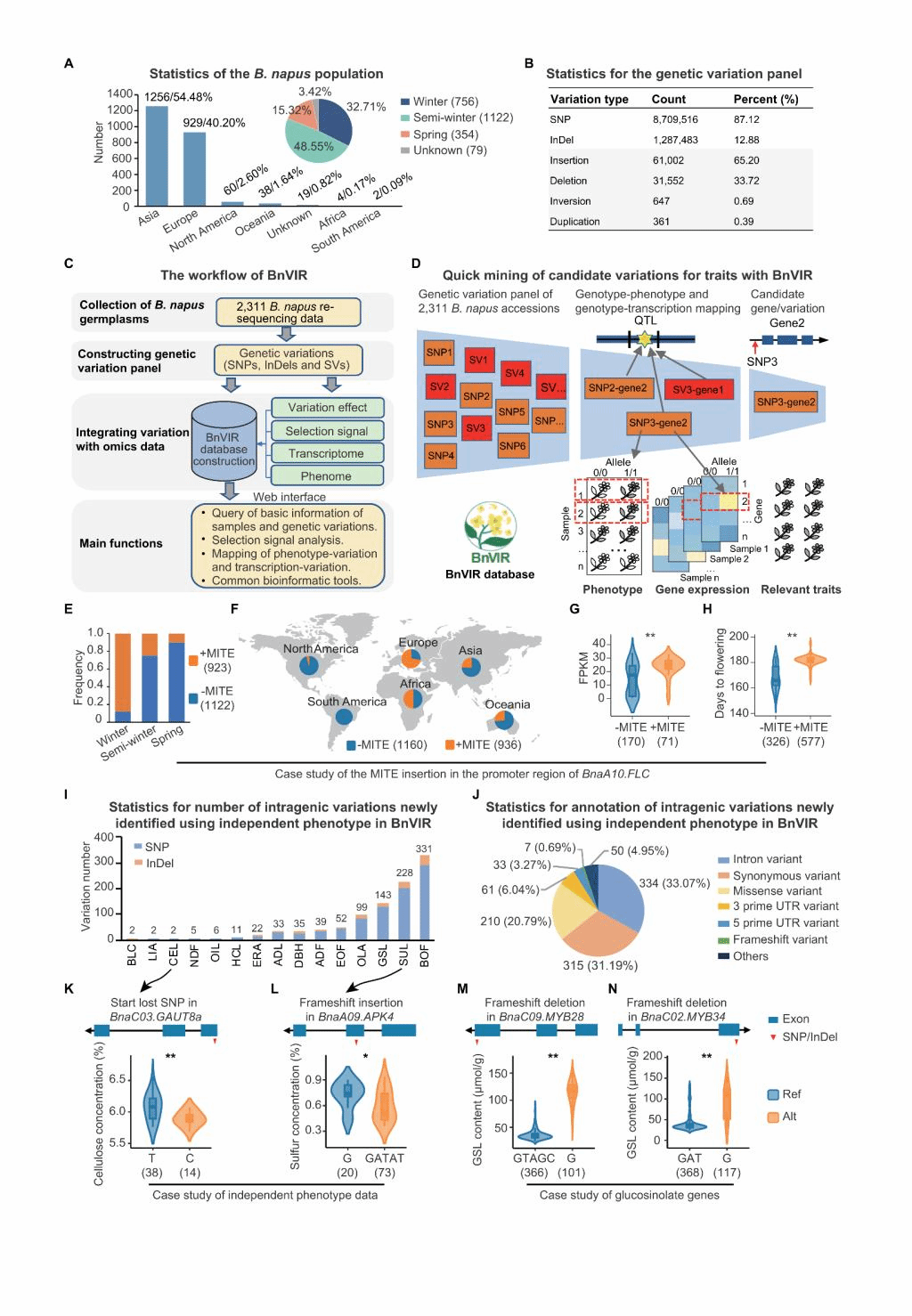
图3 使用BnVIR数据库验证/挖掘候选基因/变异的实例
(图文|杨植全 审核|杨庆勇)2月8日,我校生物信息团队杨庆勇教授课题组在国际学术期刊Molecular Plant在线发表题为“BnVIR: bridging the genotype-phenotype gap to accelerate mining of candidate variations for traits in Brassica napus”的研究论文(图1),建立了甘蓝型油菜基因型与基因表达、表型的关联,为快速、高效地挖掘油菜候选变异/基因提供了平台。该研究首先构建了一套由2,311个甘蓝型油菜核心种质鉴定的10,090,561个遗传变异的数据集,其中包括SNP、InDel和不同种类的SV,这是目前已发表的最为系统、最为完整的甘蓝型油菜遗传变异集合。该研究以其中1,703个材料的18个性状的表型数据和309个材料的RNA-seq数据为基础,构建出甘蓝型油菜变异信息资源数据库BnVIR(http://yanglab.hzau.edu.cn/BnVIR)(图2)。该数据库提供了群体水平的遗传变异基因型与表型和基因表达量的关联,可辅助快速挖掘与油菜性状相关的候选变异和基因(图3)。此外,该数据库为甘蓝型油菜种质资源的查询、群体遗传学和进化分析以及关联分析等提供了多种工具。最后,该研究通过挖掘一些已报道的(如FLC.A10、FAE1.A08、FAE1.C03、FAD2.A05)和新鉴定的候选基因和功能变异的案例分析,以及独立表型数据集的检验分析等系列工作,系统全面地展示了该数据平台在挖掘与表型相关的候选变异和基因的潜力、可靠性与稳健性(图3)。与现有甘蓝型油菜数据库不同,BnVIR提供了更加全面、更加丰富的变异及其与多个组学数据的关联和更加友好和丰富的用户界面,可显著提高研究者挖掘候选变异和基因的效率,并为甘蓝型油菜的分子育种开发提供有价值的标记。
遗传变异是物种表型多样性形成的物质基础,包括单核苷酸多态性(SNP),小片段的插入/缺失(InDel)以及大规模结构变异(SV)。目前,遗传变异已被广泛应用于人类疾病、动植物重要农艺或经济性状相关遗传位点的鉴定、重要功能基因的克隆、分子标记辅助选择育种等工作。对于作物遗传育种,准确了解遗传变异对基因的调控效应以及与表型分化的关联,对种质资源的精准鉴定和优异等位基因的挖掘具有重要作用。
油菜作为世界四大油料作物之一,在全球范围内为人类的生活和生产提供优质的食用植物油和饲料蛋白来源。我国年均油菜生产和消费居世界首位,常年种植面积1亿亩左右,占油料作物面积的50%以上,油菜安全生产在保障我国食用油供应和蛋白饲料需求上发挥着重要作用。近年来,经过一代又一代油菜科学家的努力,我国油菜研究实现了从理论、技术、产品到转化的链式创新,推动了我国油菜基础研究与应用步入世界第一方阵(《中国农业农村科技发展报告(2012—2017)》)。然而,当前我国油菜大多仍以常规育种与分子标记辅助选择育种技术为主,与国外大规模应用的以基因组选择为核心的分子育种技术相比,在品种育成速度、成功率及效率等方面仍有较大差距。因此,如何将基础研究中长期积累的资源和多组学数据(基因组、转录组等)优势转化成育种优势,是当前油菜遗传育种的核心研究内容之一。
随着后基因组代的到来,作物科学领域已积累海量数据,利用生物信息技术将大规模的多组学数据进行系统的整合和分析,并构建更友好的图形化、可视化数据库,将极大促进重要农艺性状的功能基因的发掘及机制解析,推动优异种质资源的开发和利用,加快品种选育。这对发展高效、精准的新一代生物育种,打赢种业翻身仗具有重要意义。该研究不仅为甘蓝型油菜今后的遗传育种提供了重要平台,也为其他作物进行整合和利用多组学数据推动育种产业发展提供重要参考。
我校博中bz会员登录杨庆勇教授为论文通讯作者,博中bz会员登录已毕业博士生杨植全(现广州大学博士后)、博士研究生梁聪园和硕士研究生魏璐露为论文共同第一作者。我校作物遗传改良国家重点实验室周永明教授、郭亮教授对本研究提供了重要指导。该研究得到国家自然科学基金(32070559)、国家重点研究发展计划(2021YFF1000100、2017YFE0104800)和中央高校基本科研业务费专项资金(2662018PY068)项目资助。
杨庆勇教授课题组近年来聚焦“油菜基因组学与分子育种”研究方向,先后与国内多个研究团队合作,通过系统整合和分析甘蓝型油菜的多组学数据,构建了首个油菜泛基因组(Nature Plants, 2020)及其数据库BnPIR(Plant Biotechnology Journal, 2021)、油菜转录组数据库BnTIR (Plant Biotechnology Journal, 2021)、油菜遗传变异数据库BnVIR(Molecular Plant, 2022)和植物遗传变异参考面板数据库Plant-ImputeDB (Nucleic Acids Research, 2021),搭建了我国自主可控的油菜参考基因组生态。目前在Molecular Plant、Nature Plants、Nucleic Acids Research、Plant Biotechnology Journal等杂志发表多篇油菜基因组和数据库论文。
论文链接:https://www.cell.com/molecular-plant/fulltext/S1674205222000508
英文摘要:
Background
Understanding the effect of genetic variations on traits is critical for uncovering the genetic architecture of various phenotypes. As an important oil crop, Brassica napus (B. napus) provides high-quality vegetable oil and feed protein worldwide. Substantial genotypic and phenotypic data of B. napus have become available in public databases recently, which provide important resources for genetic breeding studies of B. napus. However, the mining of candidate variations/genes has been largely hindered by the lack of solid association between genotype and phenotype and the corresponding platform.
Results
To bridge the gap between genotype and phenotype, we first constructed a large panel of 10,090,561 genetic variations from 2,311 B. napus accessions, and presented the most systematic and complete variation map of B.napus, including SNPs, InDels and various SVs. Then, phenotype data from 18 traits of 1,703 accessions and RNA-seq data of 309 accessions were used as a basis to assemble a B. napus variation information resource (BnVIR; http://yanglab.hzau.edu.cn/BnVIR). The database offers various tools for population genetic or evolutionary analysis as well as identification of variations, genes and chromosome features. Case studies of well-known functional variants (FLC.A10, FAE1.A08, FAE1.C03, FAD2.A05) and mining novel candidate variations of traits using our database as well as the independent phenotypic dataset demonstrated the power of BnVIR in mining well-known and novel candidate variations/genes of traits.
Conclusions
Different from the existing B. napus databases, BnVIR provides association between variations and phenotypes, gene expression and selection signals as well as more friendly and rich user interface. The database improves the efficiency of mining candidate variations and genes of traits and developing valuable markers for molecular breeding in B. napus.
参考文献:
Gao, Y., Yang, Z., Yang, W., Yang, Y., Gong, J., Yang, Q.Y., and Niu, X. (2021). Plant-ImputeDB: an integrated multiple plant reference panel database for genotype imputation. Nucleic Acids Res 49:D1480-D1488.
Liu, D., Yu, L., Wei, L., Yu, P., Wang, J., Zhao, H., Zhang, Y., Zhang, S., Yang, Z., Chen, G., et al. (2021). BnTIR: an online transcriptome platform for exploring RNA-seq libraries for oil crop Brassica napus. Plant Biotechnol J. 19:1895-1897.
Song, J.M., Guan, Z., Hu, J., Guo, C., Yang, Z., Wang, S., Liu, D., Wang, B., Lu, S., Zhou, R., et al. (2020). Eight high-quality genomes reveal pan-genome architecture and ecotype differentiation of Brassica napus. Nat. Plants 6:34-45.
Song, J.M., Liu, D.X., Xie, W.Z., Yang, Z., Guo, L., Liu, K., Yang, Q.Y., and Chen, L.L. (2021). BnPIR: Brassica napus pan-genome information resource for 1689 accessions. Plant Biotechnol J 19:412-414.
